細胞の輝度を測定した後、細胞の周辺領域 (細胞を取り囲む円環の領域、バックグラウンド) の輝度を測定してバックグラウンド値とし、それを細胞の輝度から減算する場合、次のような処理を行ないます。
- 画像 (下図の例では"Cells01.tif") を開き、"Measure" (測定) メニューの "Count/Size"
(カウント/サイズ) を実行して "Count/Size" (カウント/イズ) ダイアログを開きます。
- "Count/Size" (カウント/サイズ) ダイアログの "Measure" (測定) メニューから "Select
Measurements" (測定項目) を実行して "Density (mean)" [濃度(平均)]、"Density
(sum)" [濃度(合計)] 等の測定項目を選択して"OK"をクリックします。
- "Count/Size" (カウント/サイズ) ダイアログの "Manual" (手動抽出) オプションを選択してから
"Select Range" (レンジを選択) をクリックして "Segmentation" (色抽出) ウィンドウを開き、画像内の細胞の領域が赤く表示されるように画像を2値化します。
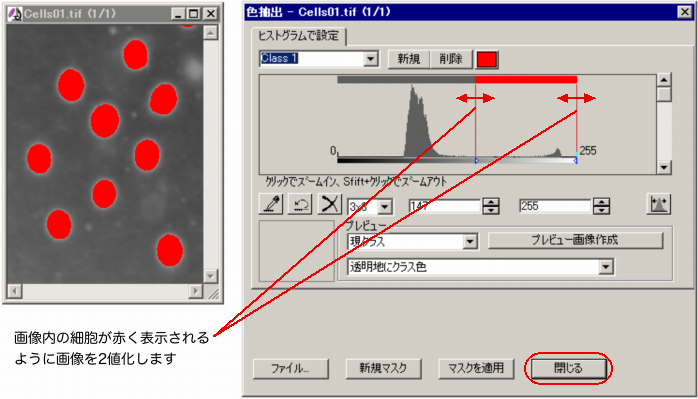
設定が終了したら、"Close" (閉じる) をクリックして "Count/Size" (カウント/サイズ) に戻ります。
- "Count" (カウント) ボタンをクリックして粒子の輝度を測定します。
- "Count/Size" (カウント/サイズ) ダイアログの "Edit" (編集) メニューにある "Delete Hidden
(Off) Objects" (除外されたオブジェクトを削除) コマンドを実行し、確認のメッセージで "Yes" (はい)
をクリックします。
- "Count/Size" (カウント/サイズ) ダイアログの "File" (ファイル) メニューにある "Data to
File " (データをファイルに保存) コマンドなどで測定結果を保存しておきます。
- "Count/Size" (カウント/サイズ) の "Image" (画像) メニューにある "Make Mask"
(マスクを作成) コマンドで、測定した範囲(=細胞の領域)を白く抜いたマスク画像 (図の例では"新規001") を作成します。
- Image-Pro Plus本体の "Edit" (編集) メニューにある "Duplicate/Crop
to AOI" (複製/AOIの切り出し) コマンドを実行して、マスク画像の複製 (図の例では"新規002") を作成します。
- "Process" (処理) メニューの "Filters" (フィルタ) を実行して "Filters" (フィルタ)
ダイアログを開き、"Morphological" (モフォロジカル) タブの "Dilate" (膨張) フィルタを適用して、細胞の領域を膨張させます。
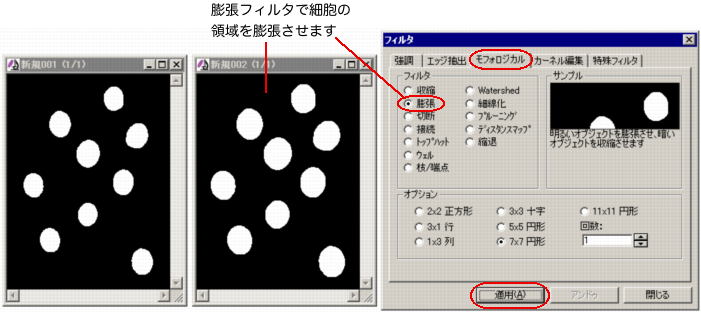
注記: 細胞の周辺領域をどの程度大きくとるかによって、"Dilate" (膨張) フィルタのオプションを設定する必要があります
[上図の例では "7x7 Circle" (7x7円形)、"Passes" (回数) = 1で処理しています]。
また、膨張後の細胞が互い に接触しないように注意する必要があります。
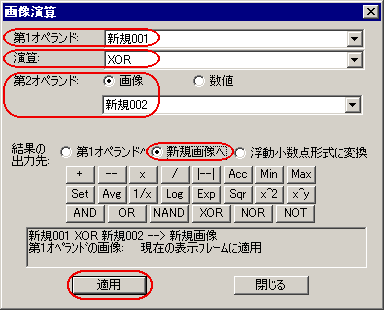
- "Process" (処理) メニューの "Operations" (演算) コマンドを実行して "Image Operations"
(画像演算) ダイアログを開き、2枚のマスク画像のXORを取ります。
|
"1st Operand" (第1オペランド) 欄で、7.の手順で作成したマスク画像
(図の例では"新規001")を選択します。
"Operation" (演算子) 欄で"XOR"を選択します。
"2nd Operand" (第2オペランド) 欄で "Image" (画像) を選択し、その下の欄で、細胞領域を膨張させたマスク画像(上の9.の手順で処理した画像="新規002")
を選択します。
"Put result in:" (結果の出力先) 欄で "New Image"
(新規画像へ) を選択します。 |
設定が終了したら、"Apply" (適用) ボタンをクリックします。
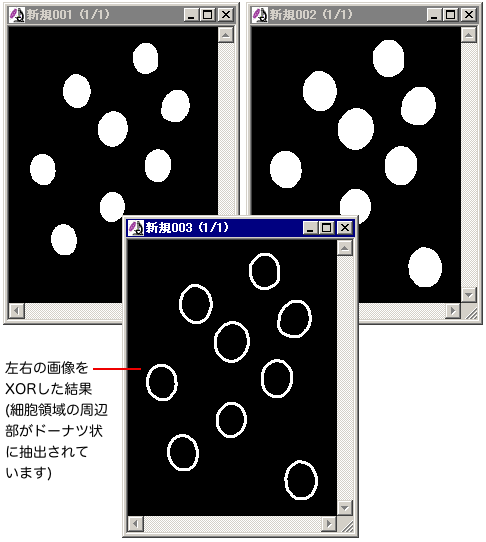
これで、元の粒子(=1番目のマスク)と膨張後の粒子(=2番目のマスク)の差となる 部分(元の粒子を取り囲むドーナツ上の領域)が新規画像(上図の例では"新規003")となって抽出されます。
- "Count/Size" (カウント/サイズ) の "Options" (オプション) ボタンをクリックして"Count/Size
Options" (カウント/サイズのオプション) ダイアログを開き、"Fill Holes" (穴を埋める) オプションを必ず非選択にしておきます。
また、"Outline Style" (アウトラインの形式) 欄で
"With Holes" (穴のアウトラインも表示する) オプションを選択します。
- "Count/Size" (カウント/サイズ) の "Automatic Bright Objects" (明るい色のオブジェクトを自動抽出)オプションを選択してから"Count"
(カウント) をクリックし、細胞を取り囲むドーナツ上の領域を抽出します。
注記:
この処理は、ドーナツ形の領域のアウトライン(輪郭)を抽出するために行ないます。ここでの測定値は不要です。
- "Count/Size" (カウント/サイズ) の "File" (ファイル) メニューから "Save Outlines"
(アウトラインを保存) を実行し、ドーナツ領域のアウトラインをファイル(.SCL)に保存します。
- 元の画像 ("Cells01.tif") をクリックして選択し、"Count/Size" (カウント/サイズ) の"File"
(ファイル) にある"Load Outlines" (アウトラインをロード) コマンドを実行して、上の14.の手順で保存した
ファイル(.SCL)を開きます。
これで元の画像にドーナツ型のアウトラインが重ね表示され、同時にその範囲内の輝度
(粒子を取り囲む円環領域の輝度)が測定されています。
- "Count/Size" (カウント/サイズ) ダイアログの "File" (ファイル) メニューにある "Data to
File " (データをファイルに保存) コマンドなどで測定結果を保存し、上の6.で保存した結果から減算し、バックグラウンド差し引き後の値を算出
します。
|